君と夏の終わり 将来の夢
大きな希望 忘れない
10年後の8月 また出会えるのを 信じて
最高の思い出を…
与你一起度过夏季的尽头 未来的梦想 庞大的希望 不会忘记
深信能够在十年后的八月重逢
成为最美好的回亿
今年四月番「あの日見た花の名前を僕達はまだ知らない。」我们仍未知道那天所看见的花的名字。这首ED至少贡献了这颗催泪弹80%的效果。据说是当年Zone乐队解散时的安可曲目,这种词,当年现场的一定哭翻了。
真TM煽情。
八月将逝。以此献给今生最后一个暑假。虽然不休假。
君と夏の終わり 将来の夢
大きな希望 忘れない
10年後の8月 また出会えるのを 信じて
最高の思い出を…
与你一起度过夏季的尽头 未来的梦想 庞大的希望 不会忘记
深信能够在十年后的八月重逢
成为最美好的回亿
今年四月番「あの日見た花の名前を僕達はまだ知らない。」我们仍未知道那天所看见的花的名字。这首ED至少贡献了这颗催泪弹80%的效果。据说是当年Zone乐队解散时的安可曲目,这种词,当年现场的一定哭翻了。
真TM煽情。
八月将逝。以此献给今生最后一个暑假。虽然不休假。
Random Forest(s),随机森林,又叫Random Trees[2][3],是一种由多棵决策树组合而成的联合预测模型,天然可以作为快速且有效的多类分类模型。如下图所示,RF中的每一棵决策树由众多split和node组成:split通过输入的test取值指引输出的走向(左或右);node为叶节点,决定单棵决策树的最终输出,在分类问题中为类属的概率分布或最大概率类属,在回归问题中为函数取值。整个RT的输出由众多决策树共同决定,argmax或者avg。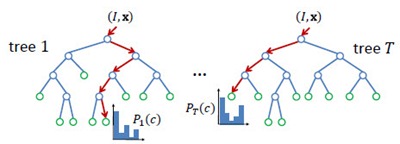
Node Test
node test通常很简单,但很多简单的拧在一起就变得无比强大,联合预测模型就是这样的东西。node test是因应用而异的。比如[1]的应用是基于深度图的人体部位识别,使用的node test是基于像素x的深度比较测试: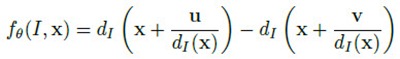
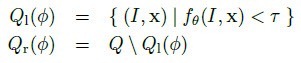
简单的说,就是比较像素x在u和v位移上的像素点的深度差是否大于某一阈值。u和v位移除以x深度值是为了让深度差与x本身的深度无关,与人体离相机的距离无关。这种node test乍一看是没有意义的,事实上也是没多少意义的,单个test的分类结果可能也只是比随机分类好那么一丁点。但就像Haar特征这种极弱的特征一样,起作用的关键在于后续的Boosting或Bagging——有效的联合可以联合的力量。
Training
RF属于Bagging类模型,因此大体训练过程和Bagging类似,关键在于样本的随机选取避免模型的overfitting问题。RF中的每棵决策树是分开训练的,彼此之间并无关联。对于每棵决策树,训练之前形成一个样本子集,在这个子集中有些样本可能出现多次,而另一些可能一次都没出现。接下去,就是循序决策树训练算法的,针对这个样本子集的单棵决策树训练。
单棵决策树的生成大致遵循以下过程:
1)随机生成样本子集;
2)分裂当前节点为左右节点,比较所有可选分裂,选取最优者;
3)重复2)直至达到最大节点深度,或当前节点分类精度达到要求。
这一过程是贪婪的。
当然对于不同的应用场合,训练过程中,会有细节上的差别,比如样本子集的生成过程、以及最优分割的定义。
在[1]中,决策树的真实样本其实是图片中的像素x,变量值则是上文提到的node test。但是,对于一张固定大小的图片而言可取的像素x是可数大量的,可取的位移(u,v)和深度差阈值几乎是不可数无限的。因此,[1]在训练单棵决策树前,要做的样本子集随机其实涉及到像素x集合的随机生成、位移(u,v)和深度差阈值组合的随机生成,最后还有训练深度图集合本身的随机生成。
最优分裂通常定义为使信息增量最大的分类,如[1]中的定义: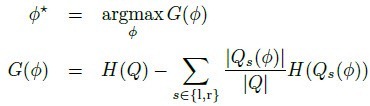
H指熵,通过分裂子集的部位标签分布计算。
源码细节:
● 训练函数
Step1:清理现场,调用clear()函数,删除和释放所有决策树,清除训练数据等;
Step2:构造适用于单棵决策树训练的参数包CvDTreeParams,主要就是对CvRTParams中一些参数的拷贝;
Step3:构建训练数据CvDTreeTrainData,主要涉及CvDTreeTrainData::set_data()函数。CvDTreeTrainData包含CvDTreeParams格式的参数包、被所有树共享的训练数据(优化结构使最优分裂更迅速)以及response类型和类数目等常用数据,还包括最终构造出来的树节点缓存等。
Step4:检查CvRTParams::nactive_vars使其不大于最大启用变量数;若nactive_vars传参为0,则默认赋值为最大启用变量数的平方根;若小于0,则报错退出;
Step5:创建并初始化一个变量活跃Mask(1×变量总数),初始化过程设置前nactive_vars个变量mask为1(活跃),其余为0(非活跃);
Step6:调用CvRTrees::grow_forest()开始生成森林。
● 生成森林
Step1:如果需要以准确率为终止条件或者需要计算变量的重要值(is_oob_or_vimportance = true),则需要创建并初始化以下数据:
oob_sample_votes 用于分类问题,样本数量×类数量,存储每个样本的测试分类;
oob_responses 用于回归问题,2×样本数量,这是一个不直接使用的数据,旨在为以下两个数据开辟空间;
oob_predictions_sum 用于回归问题,1×样本数量,存储每个样本的预测值之和;
oob_num_of_predictions 用于回归问题,1×样本数量,存储每个样本被预测的次数;
oob_samples_perm_ptr 用于存储乱序样本,样本数量×类数量;
samples_ptr / missing_ptr / true_resp_ptr 从训练数据中拷贝的样本数组、缺失Mask和真实response数组;
maximal_response response的最大绝对值。
Step2:初始化以下数据:
trees CvForestTree格式的单棵树集合,共max_ntrees棵,max_ntrees由CvDTreeParams定义;
sample_idx_mask_for_tree 存储每个样本是否参与当前树的构建,1×样本数量;
sample_idx_for_tree 存储在构建当前树时参与的样本序号,1×样本数量;
Step3:随机生成参与当前树构建的样本集(sample_idx_for_tree定义),调用CvForestTree::train()函数生成当前树,加入树集合中。CvForestTree::train()先调用CvDTreeTrainData::subsample_data()函数整理样本集,再通过调用CvForestTree::try_split_node()完成树的生成,try_split_node是一个递归函数,在分割当前节点后,会调用分割左右节点的try_split_node函数,直到准确率达到标准或者节点样本数过少;
Step4:如果需要以准确率为终止条件或者需要计算变量的重要值(is_oob_or_vimportance = true),则:
使用未参与当前树构建的样本,测试当前树的预测准确率;
若需计算变量的重要值,对于每一种变量,对每一个非参与样本,替换其该位置的变量值为另一随机样本的该变量值,再进行预测,其正确率的统计值与上一步当前树的预测准确率的差,将会累计到该变量的重要值中;
Step5:重复Step3 - 4,直到终止条件;
Step6:若需计算变量的重要值,归一化变量重要性到[0, 1]。
●训练单棵树
Step1:调用CvDtree::calc_node_value()函数:对于分类问题,计算当前节点样本中最大样本数量的类别,最为该节点的类别,同时计算更新交叉验证错误率(命名带有cv_的数据);对于回归问题,也是类似的计算当前节点样本值的均值作为该节点的值,也计算更新交叉验证错误率;
Step2:作终止条件判断:样本数量是否过少;深度是否大于最大指定深度;对于分类问题,该节点是否只有一种类别;对于回归问题,交叉验证错误率是否已达到指定精度要求。若是,则停止分裂;
Step3:若可分裂,调用CvForestTree::find_best_split()函数寻找最优分裂,首先随机当前节点的活跃变量,再使用ForestTreeBestSplitFinder完成:ForestTreeBestSplitFinder对分类或回归问题、变量是否可数,分别处理。对于每个可用变量调用相应的find函数,获得针对某一变量的最佳分裂,再在这所有最佳分裂中依照quality值寻找最最优。find函数只关描述分类问题(回归其实差不多):
CvForestTree::find_split_ord_class():可数变量,在搜寻开始前,最主要的工作是建立一个按变量值升序的样本index序列,搜寻按照这个序列进行。最优分裂的依据是![]()
也就是左右分裂所有类别中样本数量的平方 / 左右分裂的样本总数,再相加(= =还是公式看的懂些吧。。)
比如说,排序后的 A A B A B B C B C C 这样的序列,比较这样两种分裂方法:
A A B A B B | C B C C 和 A A B A B B C B | C C
第一种的quality是 (32 + 32 + 02) / 6 + (02 + 12 + 32) / 4 = 5.5
第二种的quality是 (32 + 42 + 12) / 8 + (02 + 02 + 22) / 2 = 5.25
第一种更优秀些。感性地看,第一种的左分裂只有AB,右分裂只有BC,那么可能再来一次分裂就能完全分辨;而第二种虽然右分裂只有C,但是左分裂一团糟,其实完全没做什么事情。
最优搜寻过程中会跳过一些相差很小的以及不活跃的变量值,主要是为了避免在连续变量取值段出现分裂,这在真实预测中会降低树的鲁棒性。
CvForestTree::find_split_cat_class():不可数变量,分裂quality的计算与可数情况相似,不同的是分类的标准,不再是阈值对数值的左右划分,而是对变量取值的子集划分,比如将a b c d e五种可取变量值分为{a} + {b, c, d, e}、{a, b} + {c, d, e}等多种形式比较quality。统计的是左右分裂每个类别取该分裂子集中的变量值的样本数量的平方 / 左右分裂的样本总数,再相加。同样,搜寻会跳过样本数量很少的以及不活跃的分类取值。
Step4:若不存在最优分裂或者无法分裂,则释放相关数据后返回;否则,处理代理分裂、分割左右分裂数据、调用左右后续分裂。
OpenCV2.3中Random Trees(R.T.)的继承结构:
API:
| CvRTParams | 定义R.T.训练用参数,CvDTreeParams的扩展子类,但并不用到CvDTreeParams(单一决策树)所需的所有参数。比如说,R.T.通常不需要剪枝,因此剪枝参数就不被用到。 max_depth 单棵树所可能达到的最大深度 min_sample_count 树节点持续分裂的最小样本数量,也就是说,小于这个数节点就不持续分裂,变成叶子了 regression_accuracy 回归树的终止条件,如果所有节点的精度都达到要求就停止 use_surrogates 是否使用代理分裂。通常都是false,在有缺损数据或计算变量重要性的场合为true,比如,变量是色彩,而图片中有一部分区域因为光照是全黑的 max_categories 将所有可能取值聚类到有限类,以保证计算速度。树会以次优分裂(suboptimal split)的形式生长。只对2种取值以上的树有意义 priors 优先级设置,设定某些你尤其关心的类或值,使训练过程更关注它们的分类或回归精度。通常不设置 calc_var_importance 设置是否需要获取变量的重要值,一般设置true nactive_vars 树的每个节点随机选择变量的数量,根据这些变量寻找最佳分裂。如果设置0值,则自动取变量总和的平方根 max_num_of_trees_in_the_forest R.T.中可能存在的树的最大数量 forest_accuracy 准确率(作为终止条件) termcrit_type 终止条件设置 -- CV_TERMCRIT_ITER 以树的数目为终止条件,max_num_of_trees_in_the_forest生效 -- CV_TERMCRIT_EPS 以准确率为终止条件,forest_accuracy生效 -- CV_TERMCRIT_ITER | CV_TERMCRIT_EPS 两者同时作为终止条件 |
| CvRTrees::train | 训练R.T. return bool 训练是否成功 train_data 训练数据:样本(一个样本由固定数量的多个变量定义),以Mat的形式存储,以列或行排列,必须是CV_32FC1格式 tflag trainData的排列结构 -- CV_ROW_SAMPLE 行排列 -- CV_COL_SAMPLE 列排列 responses 训练数据:样本的值(输出),以一维Mat的形式存储,对应trainData,必须是CV_32FC1或CV_32SC1格式。对于分类问题,responses是类标签;对于回归问题,responses是需要逼近的函数取值 var_idx 定义感兴趣的变量,变量中的某些,传null表示全部 sample_idx 定义感兴趣的样本,样本中的某些,传null表示全部 var_type 定义responses的类型 -- CV_VAR_CATEGORICAL 分类标签 -- CV_VAR_ORDERED(CV_VAR_NUMERICAL)数值,用于回归问题 missing_mask 定义缺失数据,和train_data一样大的8位Mat params CvRTParams定义的训练参数 |
| CvRTrees::train | 训练R.T.(简短版的train函数) return bool 训练是否成功 data 训练数据:CvMLData格式,可从外部.csv格式的文件读入,内部以Mat形式存储,也是类似的value / responses / missing mask。 params CvRTParams定义的训练参数 |
| CvRTrees:predict | 对一组输入样本进行预测(分类或回归) return double 预测结果 sample 输入样本,格式同CvRTrees::train的train_data missing_mask 定义缺失数据 |
Example:
References:
[1] OpenCV 2.3 Online Documentation: http://opencv.itseez.com/modules/ml/doc/random_trees.html
[2] Random Forests, Leo Breiman and Adele Cutler: http://www.stat.berkeley.edu/users/breiman/RandomForests/cc_home.htm
[3] T. Hastie, R. Tibshirani, J. H. Friedman. The Elements of Statistical Learning. ISBN-13 978-0387952840, 2003, Springer.